TALEN基因编辑
分子生物学的基础和兴起离不开各种酶的发现和抗体的制备。没有抗体,蛋白质就玩不转,没有限制性内切酶和连接酶,就没有基因工程,如果没有耐高温酶的发现,连PCR都是件痛苦的事情,写到这里,我突然觉得做细菌也是蛮好的,因为比较有可能发现一些功能比较奇特的蛋白,而这些蛋白,一经改造,可能就是技术上的革新。
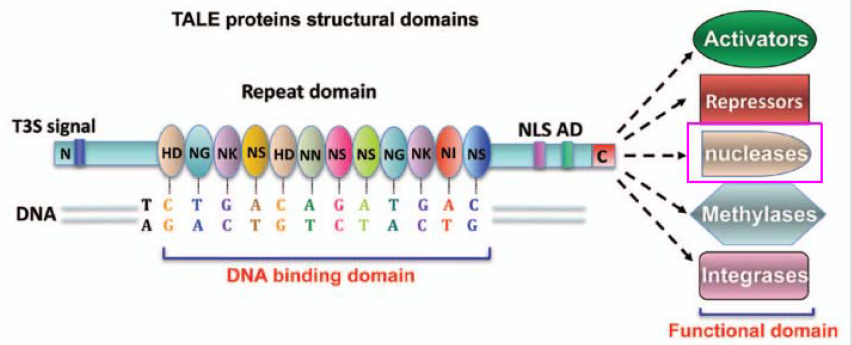
TALE(transcription activator-like effector)也不例外,最初在植物致病菌黄单胞菌(Xanthomonas)中被发现,在致病过程中,能够特异性地结合和调控植物基因。
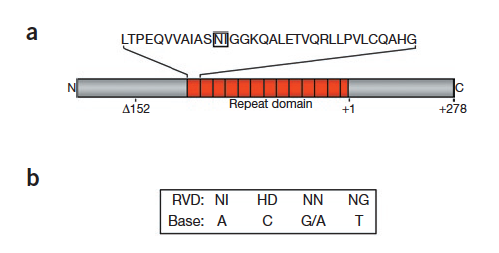
TALE的结构中间(红色部分)是重复序列,介导DNA识别,每一个重复片段为33-35个氨基酸,其中12和13位置两个氨基酸决定碱基偏好性,这两个相邻的氨基酸被称之为重复可变双残基(repeat variable di-residue, RVD),RVD所编码的靶标碱基由上图b所示,基本上你看一段TALE的重复序列,就可以预测到它将结合到什么样的DNA序列上,或者你看一段DNA序列,可以很容易地设计结合它的TALE重复序列。
N端通常是288个残基,其中Δ152做为截断点,去掉前面用于进入植物细胞的功能,而保留TALE蛋白的其它功能。C端通常是278个残基。
中间重复片段的特性使得TALE很容易被改造,应用于各种定点靶向的场景。
Activator
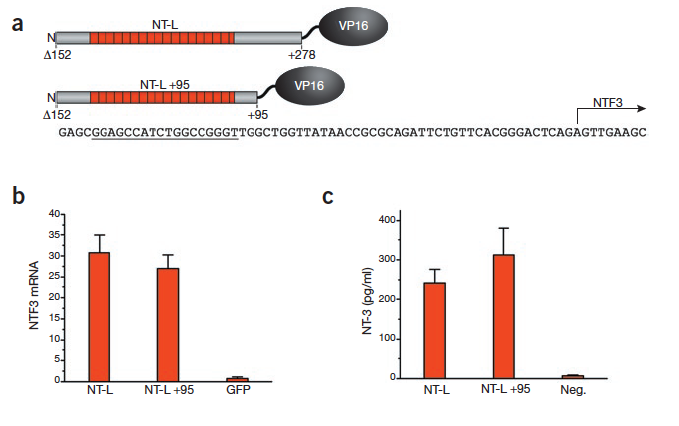
2011年发表在NBT上的文章《A TALE nuclease architecture for efficient genome editing》将TALE改造成促进特定基因表达。将天然TALE进行PCR扩增,去掉前面152个残基,重复序列改造为结合NTF3的启动子邻近区域(proximal promoter),C端连上VP16(转录激活结构域),结果诱导超过20倍的表达。NTF3基因编码一个分泌性的生长因子,对神经退行性疾病有一个的治疗功效。作者把C端截断,保留95个残基再接上VP16,同样也是mRNA超过20倍的表达。
Nuclease
位点特异性的核酸酶是基因组工程的有力工具,产生断裂的双链DNA(double strand breaks, DSBs),可以进行同源重组、靶向插入、删除。锌指蛋白(zinc finger protein)连接FokI的水解结构域,构成了锌指核酶(zinc finger nuclease, ZFN)被用来进行基因组编辑,但是ZFN的特异性和效率较差,可重复性不好。
TALE的单碱基识别能力,显然在特异性上是无可比拟的,有了TALE,TALEN(TALE-nuclease,TALE接上核酸酶)技术应运而生,特异性好,效率高,而且可以应用于各种物种。TALEN通常以同二聚体(homodimer)或异二聚体(hterodimer)对DNA从两端进行切割,两端切割点的距离(spacer length)可以是10-30bp之间,取决于linker的长度,即连接重复结构域和切割结构域的长度,长的linker,需要长的spacer,反之亦然。
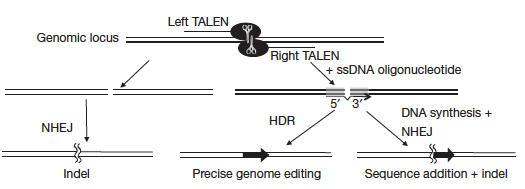
两端切割后,产生DSB,就会引起系统对其进行修复,Miller等使用TALEN对NTF3基因进行切割,产生DSB,随后进行非同源末端连接(non-homologous end joining, NHEJ)修复,NHEJ在没有同源序列做为模版的情况下进行修复,这是一个不精确的修复途径,结果产生了3-30bp的删除。
NHEJ是容易出错也不好控制的,可喜的是还存在一条精确可控的途径,同源依赖修复(homology dependent repair, HDR),在有供体ssDNA存在的情况下,通过HDR途径,可以对基因进行编辑,插入和缺失处理。
于是牛B的事情就产生了,2012年Nature的文章报道在斑马鱼活体组织里使用TALEN技术,引入了定制的EcoRV位点和修改的loxP序列。
Reference
1. Miller, Jeffrey C., Siyuan Tan, Guijuan Qiao, Kyle A. Barlow, Jianbin Wang, Danny F. Xia, Xiangdong Meng, et al. “A TALE Nuclease Architecture for Efficient Genome Editing.” Nature Biotechnology 29, no. 2 (2011): 143–148. doi:10.1038/nbt.1755. 2. Mahfouz, Magdy M, and Lixin Li. “TALE Nucleases and Next Generation GM Crops.” GM Crops 2, no. 2 (June 2011): 99–103. doi:10.4161/gmcr.2.2.17254. 3. Bedell, Victoria M., Ying Wang, Jarryd M. Campbell, Tanya L. Poshusta, Colby G. Starker, Randall G. Krug Ii, Wenfang Tan, et al. “In Vivo Genome Editing Using a High-efficiency TALEN System.” Nature (2012). doi:10.1038/nature11537.