Pubmed年度趋势
在很久很久以前,当我还在用perl的时候,有一个TGen-EUtils的perl模块,可以去爬PubMed,我喜欢用它来画所谓的pubmed趋势,比如说我在2012年的时候,就画了各个组学技术的pubmed趋势,通过每一年的文章发表量来看某个技术到底火不火,走势怎么样。
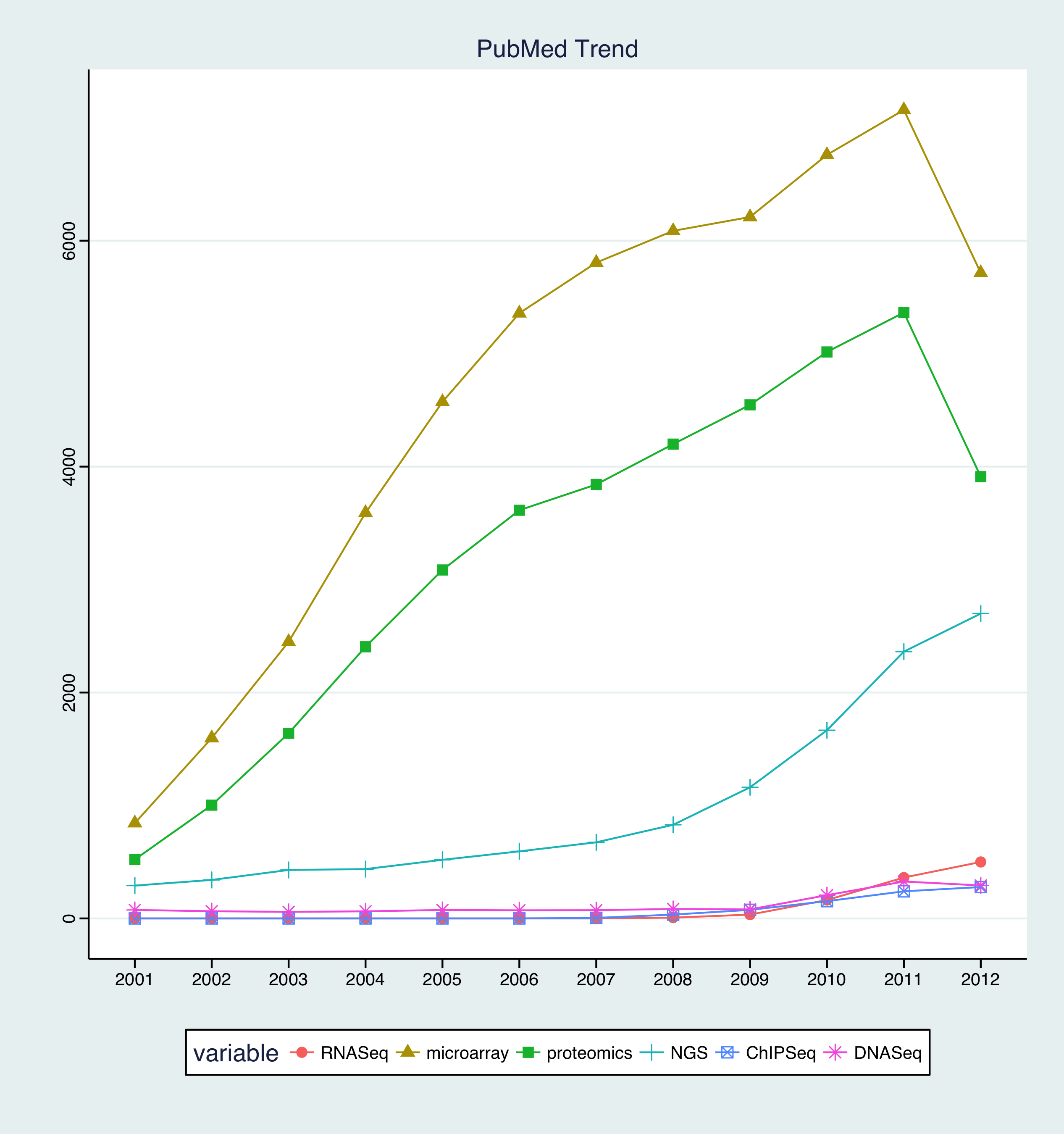
当年microarray还特别猛,但显然可以看到二代测序必须要火,也可以看到proteomics在吹了几年牛B之后,有回落的趋势。
然而这个模块,后来就找不到了,网上原来的下载地址都失效了。在我准备用RCurl去抓Pubmed数据然后用XML包来解析的时候,我发现了RISmed这个包,于是我就简单地封装了一下,又可以愉快地爬pubmed的数据了。
在2014年上public health的课的时候,每个学生要选一个病毒去介绍,我当时选了RSV,开篇我就用个pubmed趋势图来讲一下Influenza和RSV。
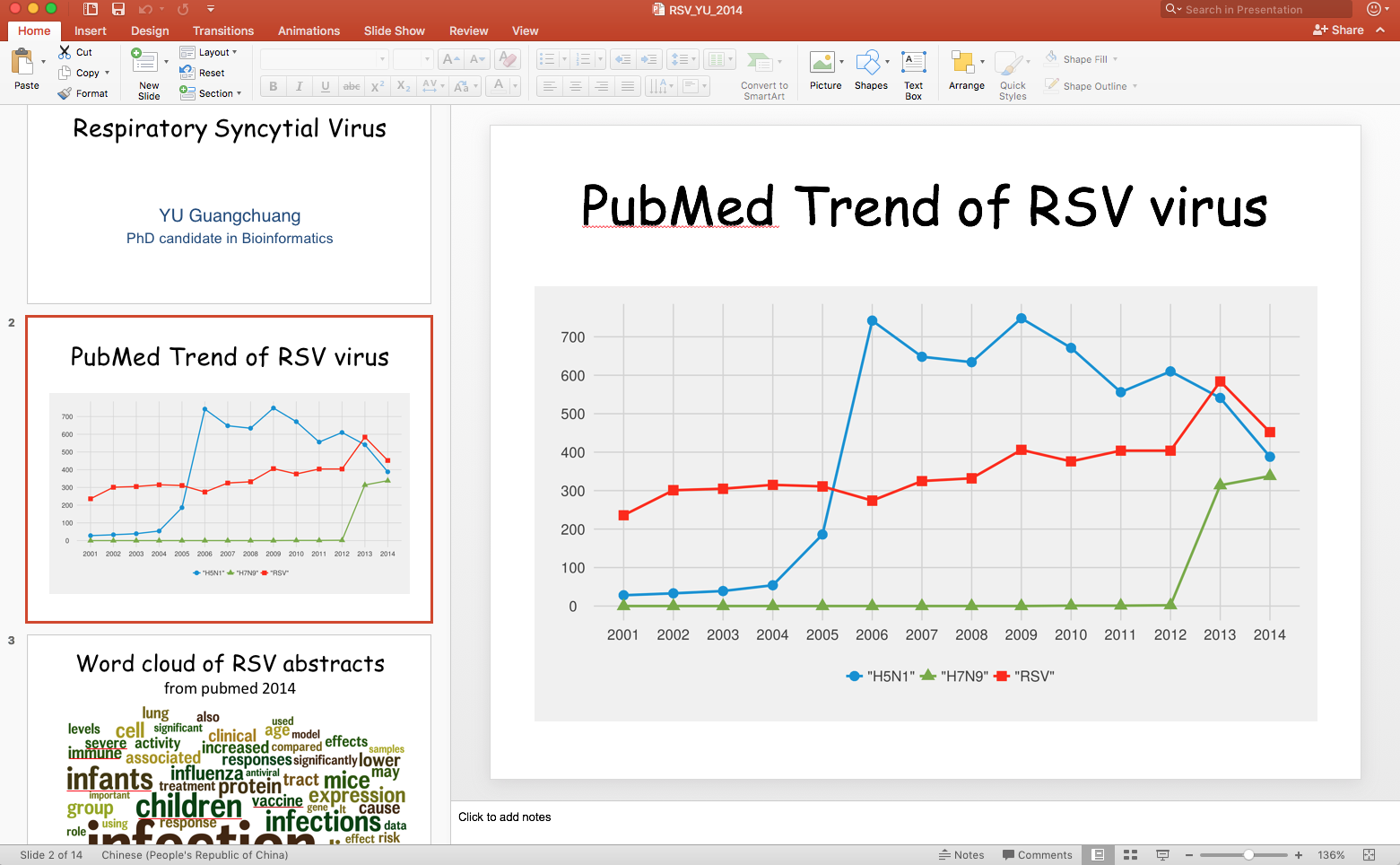
RSV的研究是很稳定的,而流感基本上是有暴发就有大量文章出现。大家都对流感有了解,看这图都知道和事实很吻合。
蓝色的线条是H5N1,2005-2006年有过暴发,2009年也同样有过暴发,在2006年之后文章量持续高涨,直到H7N9出现,而且在这个期间,两个高峰正好对应于两次暴发。

而H7N9在2013年在中国有暴发,同样文章量一下子就上去了,而且这个是亚洲的lineage,之前的文章量都是0,这是个新的高致病性病毒株。
最近我把这个函数放在了yyplot里,如果你想重复出上面这张图,很简单的代码就可以实现:(为什么search term要双引号,请参考
文档)
library(yyplot)
term <- c('"H7N9"', '"H5N1"', '"RSV"')
pm <- pubmed_trend(term, year=2001:2014)
plot(pm)
如果你想了解某个领域的研究情况和发展趋势,你可以用它来参考。比如说你老板给了几个方向,你不知道该如何选择,画个趋势出来,是不是就心里有点谱了?
当然你也可以用它来画出某个研究人员,历年来发表文章的数目,比如想看我每年发多少篇文章,下面这行代码给你答案:
pubmed_trend("Yu Guangchuang[Full Author Name]", 2010:2016)
我在博士毕业论文里,也用了一把pubmed trend:
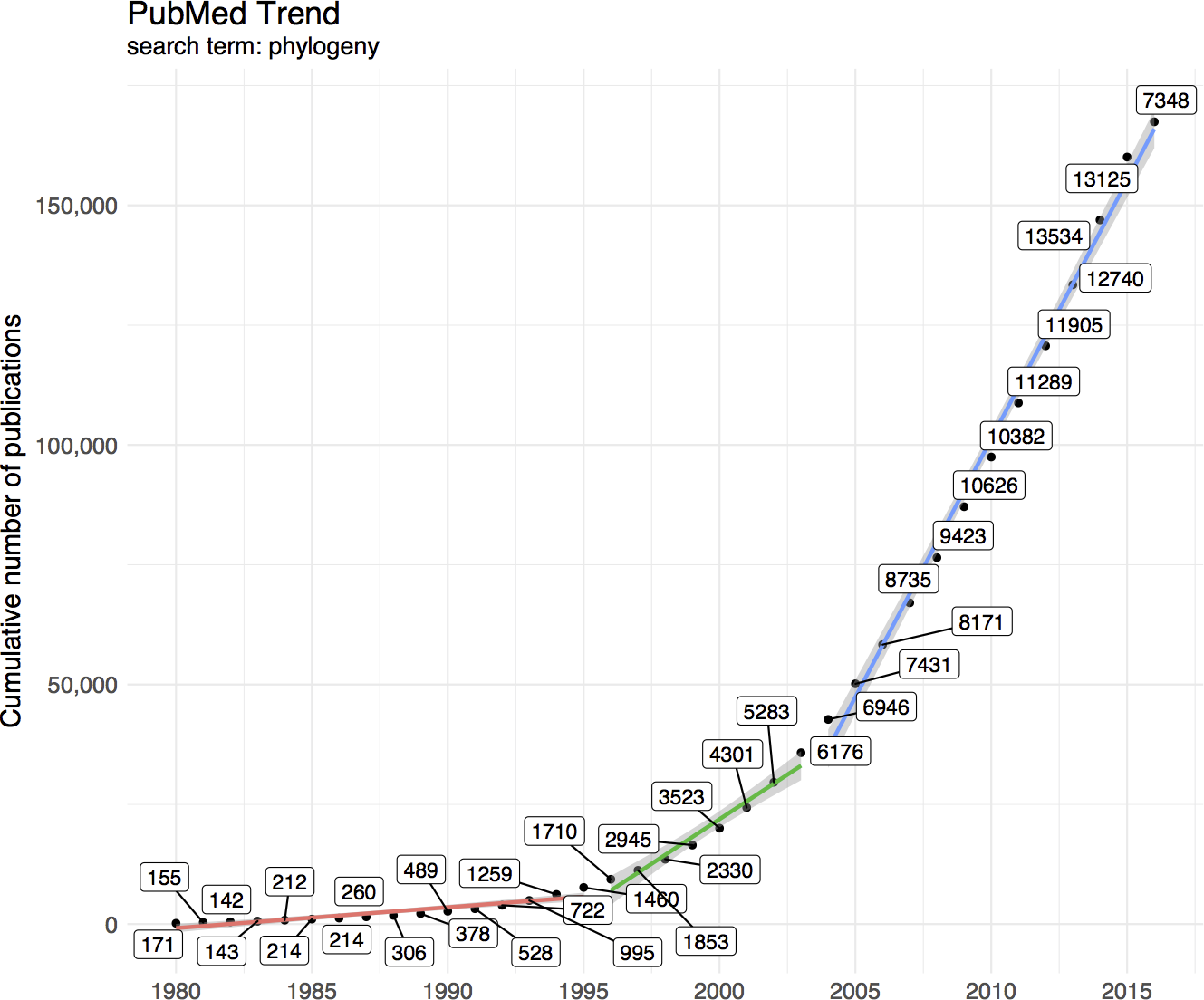
这个图我用来说明随着测序技术的发展,序列越来越多,phylogeny是研究序列的重要工具,有关phylogeny的文章也越来越多,而且我画了三段,1996年pyrosequencing测序方法提出,2004年454测序仪发布,这两个时间段都是拐点。我在写论文里可以用,你写开题报告、写基金都能用好么!