ggplot2/ggtree取子集画图
取子集对于进化树可视化来说是非常常见的,我们要区分内部节点和外部节点,我们也可能想针对某些特点的节点进行注释。
ggplot2现在所有图层都不支持直接取子集,所以呢ggtree就自己定义了一些修改的图层,包括geom_text2, geom_label2, geom_point2 和 geom_segment2,这些图层和ggplot2的版本唯一差别就是支持取子集。这样对于我们做注释来说,就更方便了。
比如说我想给内部节点打点,可以用:
ggtree(tree) + geom_text2(aes(label=label, subset=!isTip), hjust=-.2) +
geom_point2(aes(subset=!isTip), color="red", size=3)

当然ggtree有提供
geom_nodepoint图层,像上面的例子,用户不需要取子集操作,直接用geom_nodepoint即可。
ggtree所定义的这些修改版的图层,是为了进化树的注释表达力更强一些,但不单单应用于ggtree,ggplot2的用户照样受益于ggtree的图层,比如下面这个代码,产生一个散点图:
require(ggtree)
data(mpg)
p = ggplot(data = mpg, mapping = aes(x = displ, y = hwy)) +
geom_point(mapping = aes(color = class))
如果我想对其中的某些点加些文字,那么我们就需要取我们的操作的数据点的子集,再拿子集来加图层geom_text(aes_mapping, subset_of_data),这种图生物狗也常用,比如火山图,只在图上打印差异基因的名字,那么要对数据取子集。
代码如下,这里我用了subset函数取了子集,然后传给data参数。
p + geom_text(aes(label=manufacturer), data=subset(mpg, hwy > 40 | disp > 6.5))
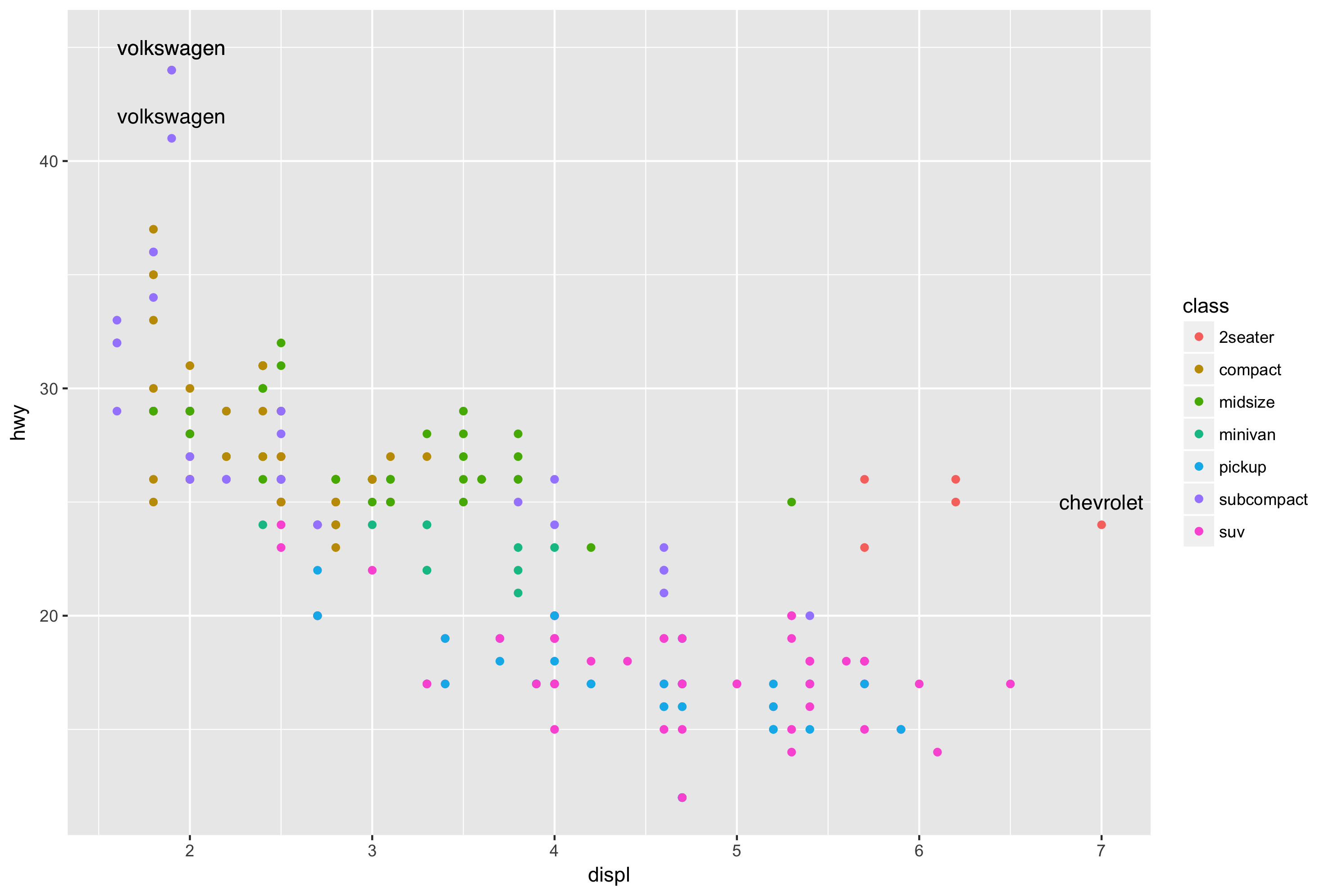
然而用ggtree定义的geom_text2图层,我们不需要传data参数,因为我们只是要取原来传给ggplot的data的子集而已,把取子集的条件放在aes映射里就行,geom_text2会去完成取子集的操作,用geom_text你需要自己显性操作数据,而用geom_text2你只需要把条件传进去即可。
下面的代码,可以产生上面一样的图:
p + geom_text2(aes(label=manufacturer, subset = hwy > 40 | displ > 6.5), nudge_y = 1)