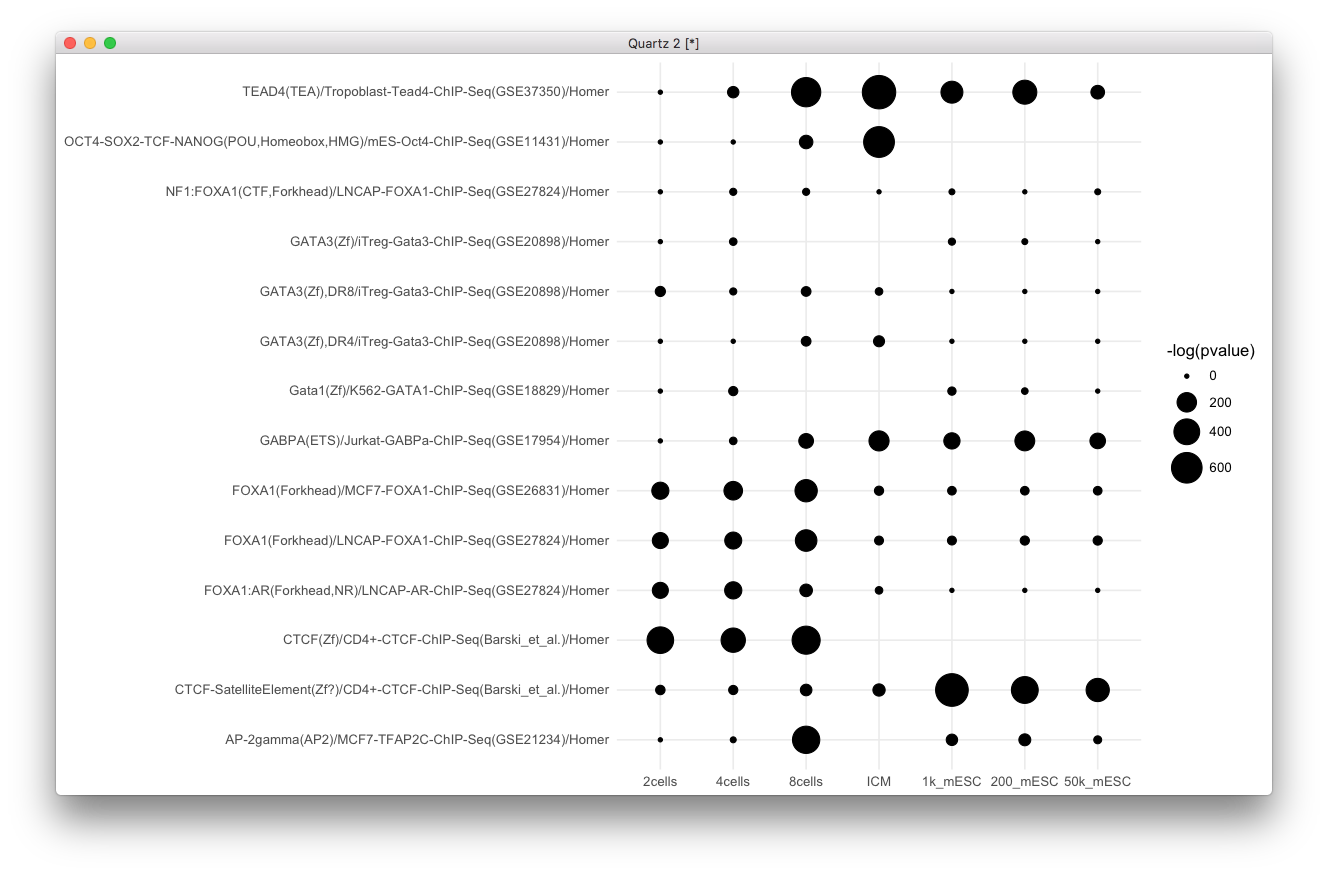
怎样用HOMER算出的P-value画出CNS级别的泡泡图?
- 第一棒:CNS里的泡泡图
- 第二棒:怎样用HOMER找motif?
- 第三棒:怎样用HOMER算出的P-value画出CNS级别的泡泡图?
这是小伙伴的命题作文,下面这个数据是曾老湿给出来的,其中header行竟然放在了最后一行,我手工把它挪到了第一行,里面还有一个数据缺失的,会导致读了数据后会有NA值,需要去除。
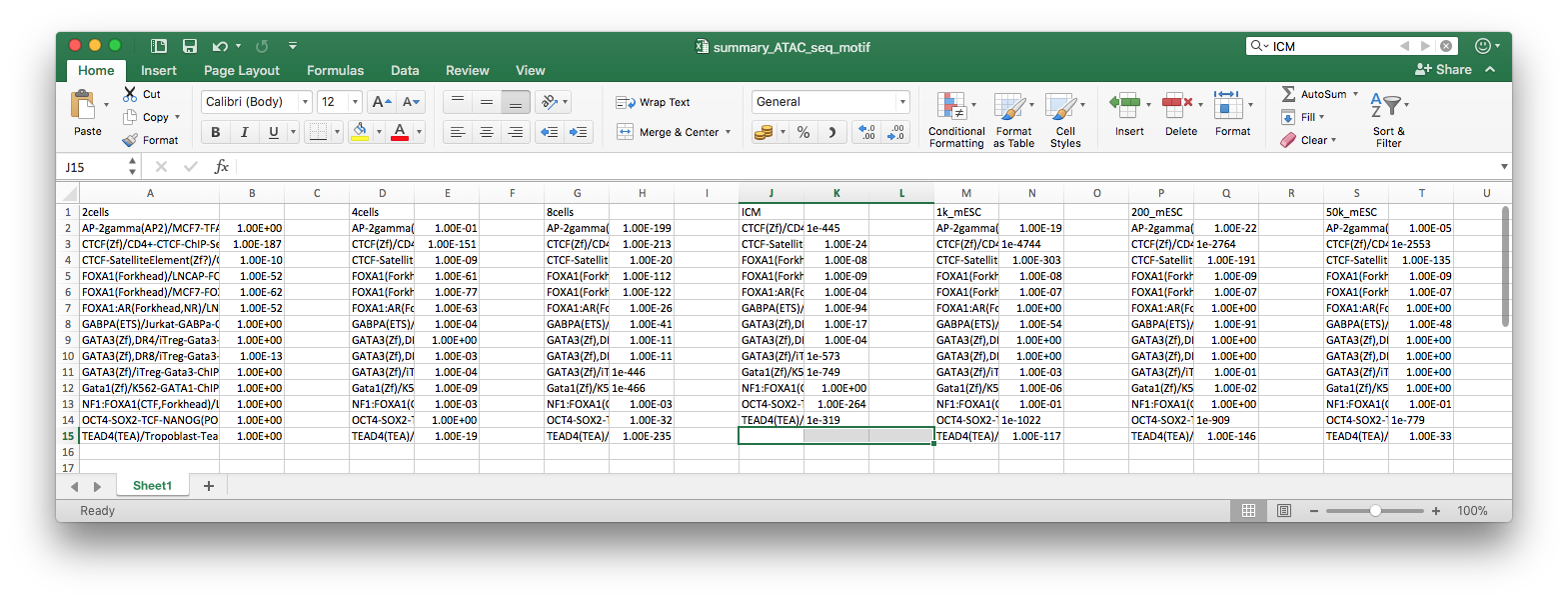
$ file summary_ATAC_seq_motif.xlsx
summary_ATAC_seq_motif.xlsx: Microsoft Excel 2007+
这是一个Excel文件,我们可以用readxl来读:
require(readxl)
x <- read_xlsx("summary_ATAC_seq_motif.xlsx")
下面是读进来的数据:
> x
# A tibble: 14 x 20
`2cells` X__1 X__2 `4cells` X__3 X__4 `8cells` X__5 X__6 ICM
<chr> <dbl> <lgl> <chr> <dbl> <lgl> <chr> <chr> <lgl> <chr>
1 AP-2gam… 1.00e+ 0 NA AP-2gam… 1.00e- 1 NA AP-2gam… 9.99… NA CTCF…
2 CTCF(Zf… 1.00e-187 NA CTCF(Zf… 1.00e-151 NA CTCF(Zf… 9.99… NA CTCF…
3 CTCF-Sa… 1.00e- 10 NA CTCF-Sa… 1.00e- 9 NA CTCF-Sa… 9.99… NA FOXA…
4 FOXA1(F… 1.00e- 52 NA FOXA1(F… 1.00e- 61 NA FOXA1(F… 9.99… NA FOXA…
5 FOXA1(F… 1.00e- 62 NA FOXA1(F… 1.00e- 77 NA FOXA1(F… 1.00… NA FOXA…
6 FOXA1:A… 1.00e- 52 NA FOXA1:A… 1.00e- 63 NA FOXA1:A… 1E-26 NA GABP…
7 GABPA(E… 1.00e+ 0 NA GABPA(E… 1.00e- 4 NA GABPA(E… 1E-41 NA GATA…
8 GATA3(Z… 1.00e+ 0 NA GATA3(Z… 1.00e+ 0 NA GATA3(Z… 9.99… NA GATA…
9 GATA3(Z… 1.00e- 13 NA GATA3(Z… 1.00e- 3 NA GATA3(Z… 9.99… NA GATA…
10 GATA3(Z… 1.00e+ 0 NA GATA3(Z… 1.00e- 4 NA GATA3(Z… 1e-4… NA Gata…
11 Gata1(Z… 1.00e+ 0 NA Gata1(Z… 1.00e- 9 NA Gata1(Z… 1e-4… NA NF1:…
12 NF1:FOX… 1.00e+ 0 NA NF1:FOX… 1.00e- 3 NA NF1:FOX… 1E-3 NA OCT4…
13 OCT4-SO… 1.00e+ 0 NA OCT4-SO… 1.00e+ 0 NA OCT4-SO… 1.00… NA TEAD…
14 TEAD4(T… 1.00e+ 0 NA TEAD4(T… 1.00e- 19 NA TEAD4(T… 9.99… NA NA
# ... with 10 more variables: X__7 <chr>, X__8 <lgl>, `1k_mESC` <chr>,
# X__9 <chr>, X__10 <lgl>, `200_mESC` <chr>, X__11 <chr>, X__12 <lgl>,
# `50k_mESC` <chr>, X__13 <chr>
因为有空白的column,所以会有NA的column出现,我们先去掉它们:
> require(purrr)
> x <- x[, map_lgl(x, function(y) !all(is.na(y)))]
> x
# A tibble: 14 x 14
`2cells` X__1 `4cells` X__3 `8cells` X__5 ICM X__7 `1k_mESC`
<chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <chr> <chr>
1 AP-2gamm… 1.00e+ 0 AP-2gamm… 1.00e- 1 AP-2gam… 9.99… CTCF… 1e-4… AP-2gamm…
2 CTCF(Zf)… 1.00e-187 CTCF(Zf)… 1.00e-151 CTCF(Zf… 9.99… CTCF… 9.99… CTCF(Zf)…
3 CTCF-Sat… 1.00e- 10 CTCF-Sat… 1.00e- 9 CTCF-Sa… 9.99… FOXA… 1E-8 CTCF-Sat…
4 FOXA1(Fo… 1.00e- 52 FOXA1(Fo… 1.00e- 61 FOXA1(F… 9.99… FOXA… 1.00… FOXA1(Fo…
5 FOXA1(Fo… 1.00e- 62 FOXA1(Fo… 1.00e- 77 FOXA1(F… 1.00… FOXA… 1E-4 FOXA1(Fo…
6 FOXA1:AR… 1.00e- 52 FOXA1:AR… 1.00e- 63 FOXA1:A… 1E-26 GABP… 9.99… FOXA1:AR…
7 GABPA(ET… 1.00e+ 0 GABPA(ET… 1.00e- 4 GABPA(E… 1E-41 GATA… 1.00… GABPA(ET…
8 GATA3(Zf… 1.00e+ 0 GATA3(Zf… 1.00e+ 0 GATA3(Z… 9.99… GATA… 1E-4 GATA3(Zf…
9 GATA3(Zf… 1.00e- 13 GATA3(Zf… 1.00e- 3 GATA3(Z… 9.99… GATA… 1e-5… GATA3(Zf…
10 GATA3(Zf… 1.00e+ 0 GATA3(Zf… 1.00e- 4 GATA3(Z… 1e-4… Gata… 1e-7… GATA3(Zf…
11 Gata1(Zf… 1.00e+ 0 Gata1(Zf… 1.00e- 9 Gata1(Z… 1e-4… NF1:… 1 Gata1(Zf…
12 NF1:FOXA… 1.00e+ 0 NF1:FOXA… 1.00e- 3 NF1:FOX… 1E-3 OCT4… 1E-2… NF1:FOXA…
13 OCT4-SOX… 1.00e+ 0 OCT4-SOX… 1.00e+ 0 OCT4-SO… 1.00… TEAD… 1e-3… OCT4-SOX…
14 TEAD4(TE… 1.00e+ 0 TEAD4(TE… 1.00e- 19 TEAD4(T… 9.99… NA NA TEAD4(TE…
# ... with 5 more variables: X__9 <chr>, `200_mESC` <chr>, X__11 <chr>,
# `50k_mESC` <chr>, X__13 <chr>
现在就OK了,那么这个数据长这样子,一列是motif,紧跟着一列是p值,而motif列的名字是相应的实验条件,这些信息我们都是要的。
y <- map_df(seq(1, length(x), 2), function(i) {
res <- x[,i:(i+1)]
names(res) <- c("motif", "pvalue")
res[[2]] %<>% as.numeric
res$type = names(x)[i]
res <- res[!is.na(res$motif),]
res
})
y$type = factor(y$type, levels=names(x)[seq(1, length(x), 2)])
实验条件存在type这个列里,设置了level,这样按照原本excel里的顺序来。现在的数据长这样子:
> y
# A tibble: 97 x 3
motif pvalue type
<chr> <dbl> <fct>
1 AP-2gamma(AP2)/MCF7-TFAP2C-ChIP-Seq(GSE21234)/Homer 1.00e+ 0 2cel…
2 CTCF(Zf)/CD4+-CTCF-ChIP-Seq(Barski_et_al.)/Homer 1.00e-187 2cel…
3 CTCF-SatelliteElement(Zf?)/CD4+-CTCF-ChIP-Seq(Barski_et_al.… 1.00e- 10 2cel…
4 FOXA1(Forkhead)/LNCAP-FOXA1-ChIP-Seq(GSE27824)/Homer 1.00e- 52 2cel…
5 FOXA1(Forkhead)/MCF7-FOXA1-ChIP-Seq(GSE26831)/Homer 1.00e- 62 2cel…
6 FOXA1:AR(Forkhead,NR)/LNCAP-AR-ChIP-Seq(GSE27824)/Homer 1.00e- 52 2cel…
7 GABPA(ETS)/Jurkat-GABPa-ChIP-Seq(GSE17954)/Homer 1.00e+ 0 2cel…
8 GATA3(Zf),DR4/iTreg-Gata3-ChIP-Seq(GSE20898)/Homer 1.00e+ 0 2cel…
9 GATA3(Zf),DR8/iTreg-Gata3-ChIP-Seq(GSE20898)/Homer 1.00e- 13 2cel…
10 GATA3(Zf)/iTreg-Gata3-ChIP-Seq(GSE20898)/Homer 1.00e+ 0 2cel…
# ... with 87 more rows
那么画图就是显而易见的事情了,这里应该指出一点是曾老湿没有给我相应FPKM值,如果有表达值的话,与上面的y,full_join一下,然后就可以aes(color=FPKM)给点填充颜色了,这里用到了《搞大你的点,让我们画真正的气泡图》所教给大家的招数,如果你觉得Motif名字太长,当然你可以把无用的信息删除掉,像这个例子中都有Homer的字样,可以删的,从画图的角度来说,可以强制换行,这一招,我也是教给大家的,请看《wrapping labels in ggplot2》,另外你可能还给motif分类,像给motif名字上不同的颜色,这个算是容易的,但想生成图例,却是一件比较tricky的事情,而我也是教给大家的,请移步《映射变量给axis上色》,似乎没有我没讲过的疑难杂症。
library(ggplot2)
ggplot(y, aes(type, motif)) +
geom_point(aes(size=-log(pvalue))) +
theme_minimal() +
xlab(NULL) + ylab(NULL) +
scale_size_continuous(range=c(1, 10))