dotplot支持使用formula指定x轴变量
使用barplot来展示富集分析结果是很常用的,而dotplot比较barplot来说,多了一个点大小的信息,可以比barplot展示多一个信息,所以是比较推荐的,我之前已经写了《dotplot展示富集分析结果》和《dotplot for GSEA》两篇文章,dotplot虽然简单,很多人会觉得会容易用ggplot2画出来,但其实有些细节,比如《为什么画出来的点比指定的数目要多?》,有些技巧,比如《搞大你的点,让我们画真正的气泡图》,是很多新手所不具备的,图虽然简单,但老司机的飚车技能也不可小看哦,所以我在《听说你也在画dotplot,但是我不服!》的文后就说了一句话:
clusterProfiler之所以好,因为真的考虑了很多细节!请放开那图,让clusterProfiler来画!
在对于细节上的追求,是永远没有止境的,下一个版本的dotplot,会使用我的《enrichplot: 让你们对clusterProfiler系列包无法自拔》,这一版本也有新功能加入哦,还是那句话,图虽简单,但细节是可以不断打磨的,给用户更好的能力、更大的自由度去呈现结果。
下一个版本的Bioconductor会在5月1号发布,如果想尝鲜,可以参考《安装enrichplot及升级clusterProfiler》进行安装。
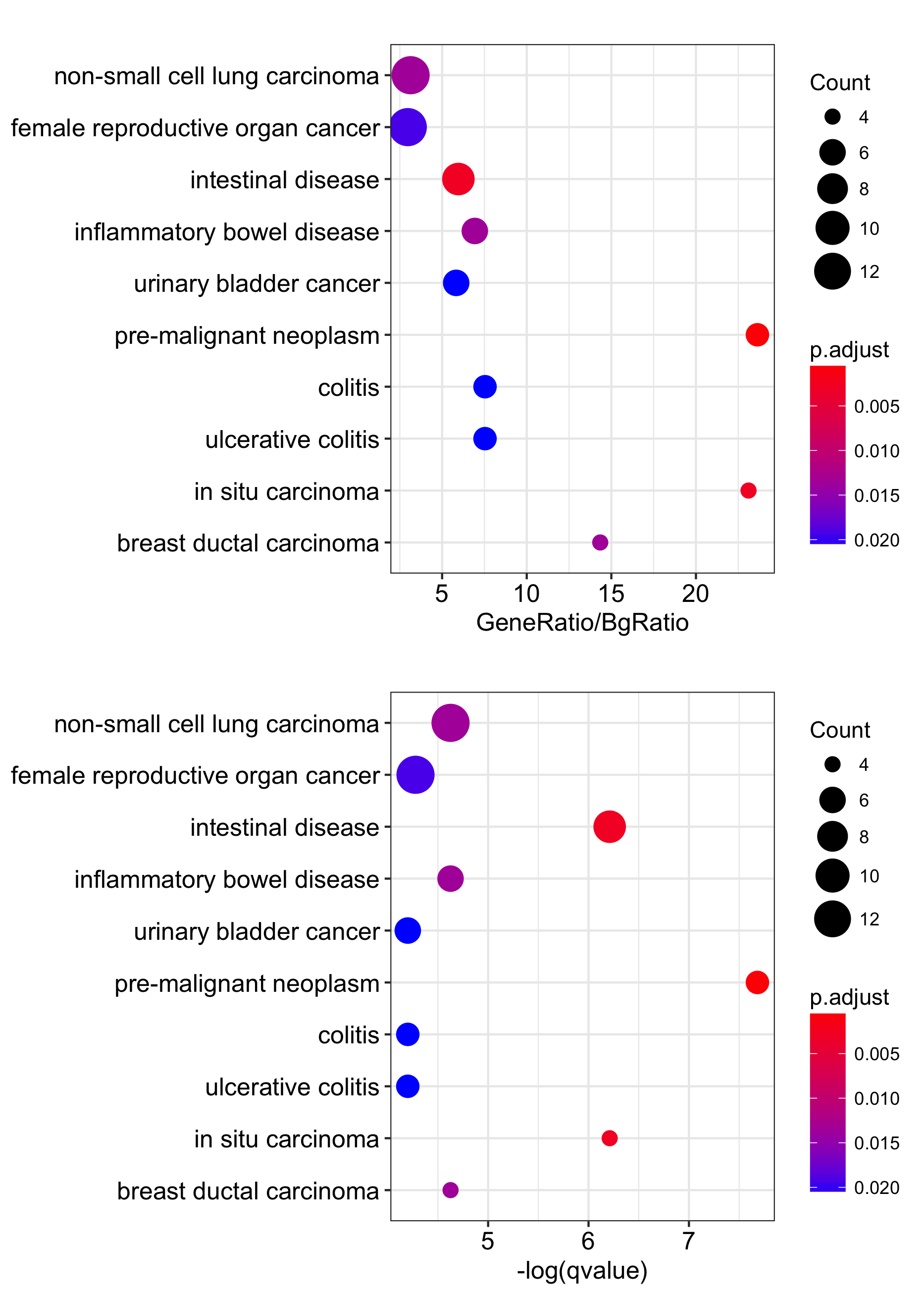
今天要介绍的新功能是对于x轴的变量,你除了指定特定的变量之外,你还可以使用formula,也就是基于(一个或多个)变量衍生的变量。比如说,你不想用GeneRatio或Count这些做为x轴,而是想要GeneRatio/BgRatio,再比如你想要用-log(p.adjust)之类的,都可以。
library(DOSE)
data(geneList)
de = names(geneList)[1:100]
res = enrichDO(de)
require(enrichplot)
dotplot(res, x = ~GeneRatio/BgRatio)
dotplot(res, x = ~ -log(qvalue))
除了上面的实例之外,你还可以试试别的。比如我在华大的报告中看到他们用RichFactor来做为x轴变量,在华大的文档中RichFactor是这样定义的:
RichFactor指差异表达的基因中位于该pathway条目的基因数目与所有有注释基因中位于该pathway条目的基因总数的 比值,RichFactor越大,表示富集的程度越大。
那么我们怎么来画呢?结果中有Count对应的就是分子,而分母是没有直接对应的变量的,但可以用BgRatio * N来得到,这里N是所有有注释的基因总数,也就是BgRatio这个变量所显示的分母,我们只要BgRatio的分子就可以了。
这里说得似乎有点绕,比如下面这个疾病描述:
ID Description GeneRatio BgRatio
DOID:0060071 DOID:0060071 pre-malignant neoplasm 5/77 22/8007
pvalue p.adjust qvalue
DOID:0060071 1.671524e-06 0.0006401937 0.0004609887
geneID
DOID:0060071 6280/6278/10232/332/4321
Count
DOID:0060071 5
RichFactor的分子是差异基因中对应的数目5,而分母是对应于这个疾病的所有有注释的基因总数22,也就是BgRatio = 22/8007的分子。
那么我们怎么用RichFactor来画图呢?请试试下面的代码,看到这里,你应该知道你可以用各种各样的衍生变量来做为x轴画图了,dotplot函数给了你更大的自由,提供更多的灵活性。
N <- 8007
dotplot(res, x = ~Count/(BgRatio * N))
富集分析、结果展示,请认准Y叔牌的clusterProfiler系列包!