old habits die hard
从2011年1月我就在实验室的QQ群里发群邮件说IPI关门,时至今日,已经关门3年了,主页上一直停留在关门大吉的那一刻。
我不断在邮件里, lab meeting上强调要换成uniprot来搜库,然而时至今日,依然还是有很多的人在使用IPI,想想真可怕,实验室真是100年不更新一下数据啊。 另外一个我非常讨厌的就是GI号,它压根就不是正儿八经的ID号,但他们从来就不愿意尝试改变。
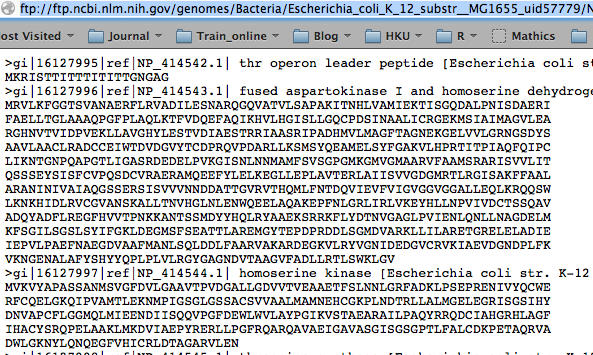
比如上面这个蛋白质序列的FASTA文件,注释行有很多信息,比如:
>gi|16128001|ref|NP_414548.1| putative transporter [Escherichia coli str. K-12 substr. MG1655]
显然搜库时可以使用NP_414548.1做为ID,这个问题我说过N多遍,但他们一定会用他们惯用的gi|16128001来做ID。
问题是很明显的:
GI number (sometimes written in lower case, “gi”) is simply a series of digits that are assigned consecutively to each sequence record processed by NCBI. The GI number bears no resemblance to the Accession number of the sequence record. The GI number has been used for many years by NCBI to track sequence histories in GenBank and the other sequence databases it maintains. The VERSION system of identifiers was adopted in February 1999 by the International Nucleotide Sequence Database Collaboration (GenBank, EMBL, and DDBJ).
这玩意不是蛋白的ID,是NCBI拿来做历史记录的,所以它老变,而且各种蛋白注释信息都不会基于GI号来。这就是问题了,给我一堆GI号,我要做分析的话,就得转成别的ID,但这货老变身。比如以蛋白NP_414680.4为例,它已经有几次revision:
>gi|90111089|ref|NP_414680.4| putative fimbrial-like adhesin protein [Escherichia coli str. K-12 substr. MG1655]
MIKTTPHKIVILMGILLSPSVFATDINVEFTATVKATTCNITLTGNNVTNDGNNNYTLRIPKMGLDKIAN
KTTESQADFKLVASGCSSGISWIDTTLTGNASSSSPKLIIPQSGDSSSTTSNIGMGFKKRTTDDATFLKP
NSAEKIRWSTDEMQPDKGLEMTVALRETDAGQGVPGNFRALATFNFIYQ
在这几次revision中,NP是不变的,但gi号做为NCBI数据库内部使用的primary ID,每一次revision都要有赋予unique的gi号,可以从http://www.ncbi.nlm.nih.gov/protein/NP_414680.4?report=girevhist看到revision的历史记录,4个version对应4个gi号。
上面的序列数据来自ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/Escherichia_coli_K_12_substr__MG1655_uid57779/NC_000913.faa,这个实验室100年不更新数据,也不知道搜的是什么时间的库,但我从NCBI上获得的数据,只有这一个最新版本,好多GI号都变了,对不上,就没办法转成诸如NP_414680.4这样的参考序列号。
当然,对于这些老黄历的GI号,还是可以转的,通过NCBI的eutils,在线去抓数据回来解析,只不过把非常简单的事情复杂化了而已。
我一直强调搜库时匹配参考序列号,像NP_414680是永远都不会变的,它就是某个蛋白的终身编号,版本更新只会改变后面的.4。但某组永远都只会给出GI号,我在多次被虐身虐心之后,对这货竟然也熟悉了起来,sigh…
2016年3月,NCBI终于决定要抛弃GI号:
NCBI is phasing out sequence GIs - use Accession.Version instead!
2016年9月之后,将不再有GI号的存在.