ChIPseq从入门到放弃
接下来要出一个ChIPseq系列,讲一讲ChIPseq和我的ChIPseeker包,从入门到放弃是我自己的个人写照。我做ChIPseq总共也就3个月的时间,做的事情并不多,在一知半解的情况下写下了ChIPseeker包。
我当时被要求做ChIPseq分析是为他人做嫁衣,而且是完全白干那种,但做为学生,白干也得干。

当时一开始使用ChIPpeakAnno做注释,但用UCSC genome browser检验结果的时候,发现对不上。在对ChIPpeakAnno包不满意的情况下,开始着手写ChIPseeker,其实在使用ChIPpeakAnno的时候,我就有写代码对结果做一些可视化,所以未有ChIPseeker先有ChIPseeker的部分可视化功能。当时写了篇博客文说ChIPpeakAnno的问题,一个月后就在Bioconductor上发表了ChIPseeker,这包完全是我半夜在宿舍里写出来的。
当时还在生物系,被我炒掉的前老板每天要求必须起码在实验室待够12小时,我每天都待到10点半左右才回宿舍,日常在实验室里啥都干不了,白天各种瞎折腾,晚上还要陪他聊天,但说来说去,每天几乎都差不多,无非是他很牛逼,我们这帮人读他phd实在太幸运,日复一日传销式洗脑。而我因为结婚了,家又离得近,周末回家,白天经常多一段单独对我的洗脑,做为一个PhD学生,在发表文章之前是不能够有周末的。每天10半从实验室里出来,回到宿舍11点,跟老婆打电话再洗澡,12点。然后从12点开始写代码到2点睡觉,才有了这个包。
虽然是一知半解的时候开发的,但还是受到大家的欢迎,半年前Matt邀请我去人大做报告时,也专门提到了ChIPseeker。
也有美国的助理教授,跟我要paper,说是上课的时候,要给学生读的,这广告效果我给满分。
文章发表了一年,已经被33篇文章引用,其中不乏有影响因子比较高的杂志:

下面是其中一些引用文章的图:
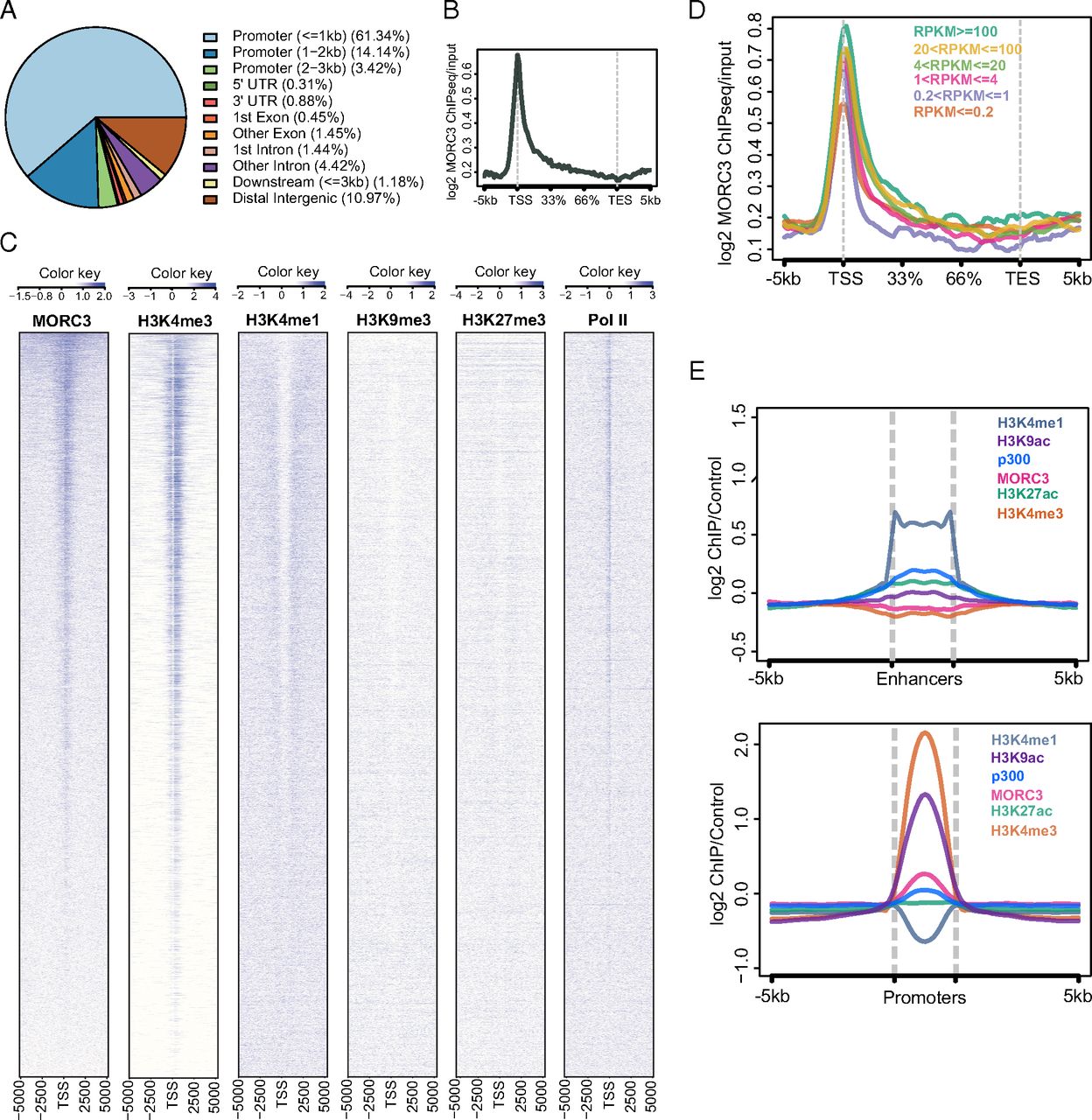
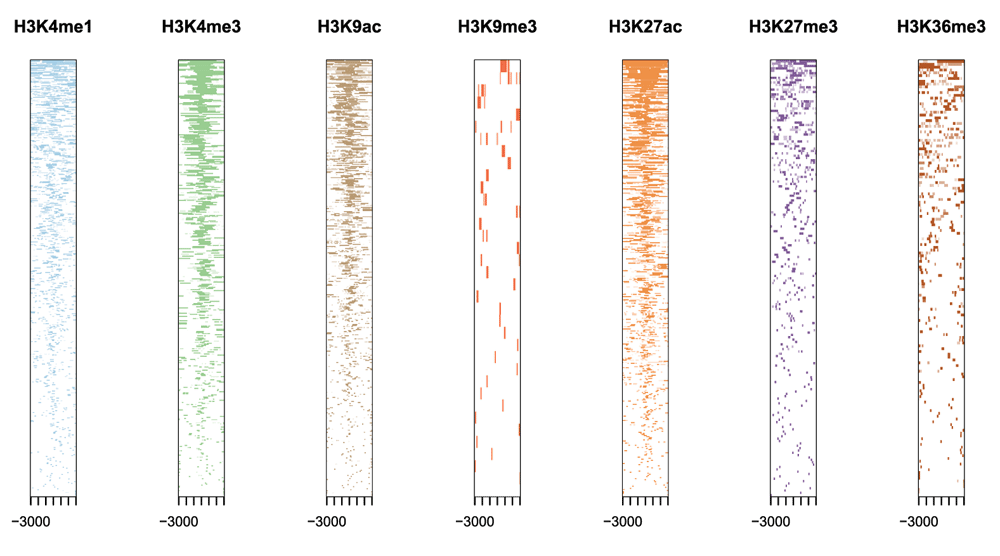
虽然ChIPseeker是我写给自己做ChIPseq注释的,但Ming Tang (https://github.com/crazyhottommy/ChIP-seq-analysis)用它去做DNA breakdown注释,当然像lincRNA注释也是有人做并且完全是支持的。有一些我以前从没在文档里提到的东西,也应该会在这个系列里写出来。
这个系列基本上是围绕着ChIPseeker的功能而来,名副其实从入门到放弃,因为我自己也是入了门然后放弃,如果想看从入门到精通的,这显然不适合你。
然而今天只是个剧透,敬请期待。
Citation
Yu G, Wang LG and He QY*. ChIPseeker: an R/Bioconductor package for ChIP peak annotation, comparison and visualization. Bioinformatics 2015, 31(14):2382-2383.