听说你的KEGG分析有大量的基因没注释
github上的问题,问了两个问题,这是其中第二个:
Meanwhile, when I fed the ENTREZID to enrichKEGG, it show me two unreasonable results:
kegg_enrich = enrichKEGG(
gene = new_ids$ENTREZID,
organism = 'hsa',
keyType = 'ncbi-geneid',
pvalueCutoff = 0.05,
#pAdjustMethod = 'BH',
#qvalueCutoff = 0.2,
use_internal_data = FALSE
)
head(kegg_enrich)
ID Description GeneRatio BgRatio pvalue p.adjust qvalue
hsa04380 hsa04380 Osteoclast differentiation 14/281 128/7299 0.0003817040 0.04767338 0.04508266
hsa04070 hsa04070 Phosphatidylinositol signaling system 12/281 99/7299 0.0003875885 0.04767338 0.04508266
I noted that only 281 genes are remained(there are 700+ genes in my list). In case that there is something wrong wtihin my gene list, I also tried my list with DAVID. It gave me reasonable results. So this is my second question, why enrichKEGG cannot recognize my geneids?
大概说他有700+的基因,clusterProfiler做KEGG时只认得281个,比DAVID要差。他有提供一个gene_list.txt在github上,这样我就有可能帮他看一下。
说比DAVID差是不可能,我把他的gene list上传到DAVID上,只认得197个,啪啪啪,DAVID的脸好疼。
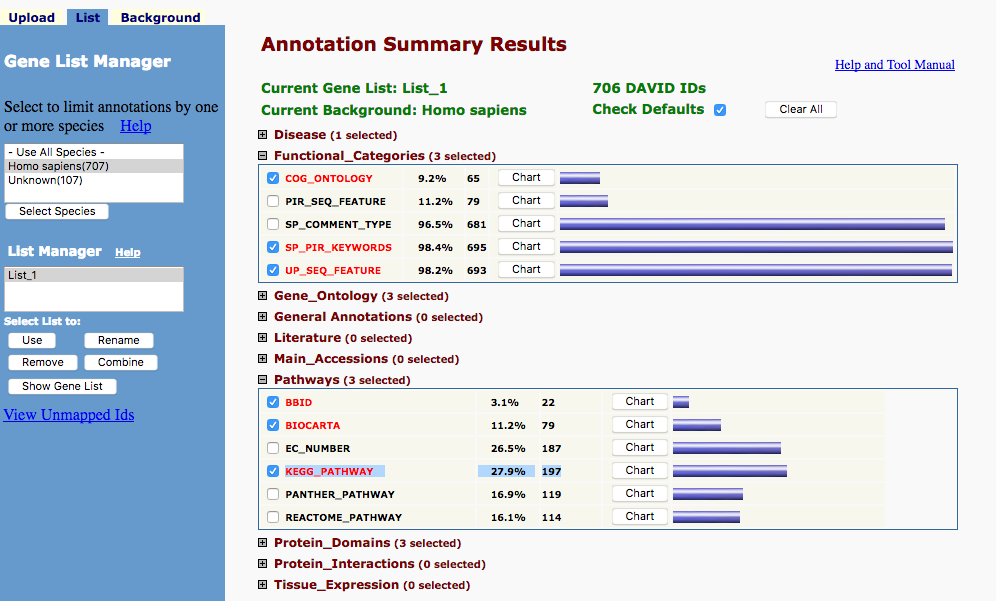
比DAVID多出42%的基因注释:
> (281-197)/197
[1] 0.4263959
那么问题回到为什么这么多基因没有注释,这大家得问KEGG,KEGG的通路注释主要集中在代谢通路上,很多基因没有注释是事实,你可以尝试一下用我的另一个包ReactomePA,试一下reactome pathway分析。
当然我不能只说「没注释就是没注释啊」,「我也很绝望啊」,我必须还给出事实。
clusterProfiler给出了很多的小工具,让我们检验结果相当之容易,我们可以把基因映射到KEGG通路上去:
> bitr_kegg(geneID = new_ids$ENTREZID,
+ fromType='ncbi-geneid', toType='Path',
+ organism='hsa') -> xx
Warning message:
In bitr_kegg(geneID = new_ids$ENTREZID, fromType = "ncbi-geneid", :
62.98% of input gene IDs are fail to map...
提示说~63%的基因是没有KEGG注释的,有注释的基因我们可以通过转换后的结果得到:
> head(xx)
ncbi-geneid Path
1 10023 hsa04310
2 10023 hsa05224
3 10023 hsa05225
4 10057 hsa01523
5 10057 hsa02010
6 10114 hsa04218
而没有注释的,也很容易通过比较而得到:
> new_ids$ENTREZ[!new_ids$ENTREZID %in%
+ xx[, "ncbi-geneid"]] %>% head
[1] "100506775" "143458" "57649" "54899" "220323" "8728"
那么有了这个结果,我们就可以去KEGG数据库去核查了,看看clusterProfiler有没有欺骗你。
KEGG的基因注释可以通过类似于下面这样的链接得到:
http://www.genome.jp/dbget-bin/www_bget?hsa:ENTREZID.
比如说,上面有注释的基因10023, 通过这个页面:
http://www.genome.jp/dbget-bin/www_bget?hsa:10023,
可以确认确实是有注释的。
而没有注释的基因100506775, 通过网页:
http:www.genome.jp/dbget-bin/www_bget?hsa:100506775,
也确实可以确认是没有KEGG注释的。
你可以多试几个,然后可以得到结论:clusterProfiler诚不欺我!