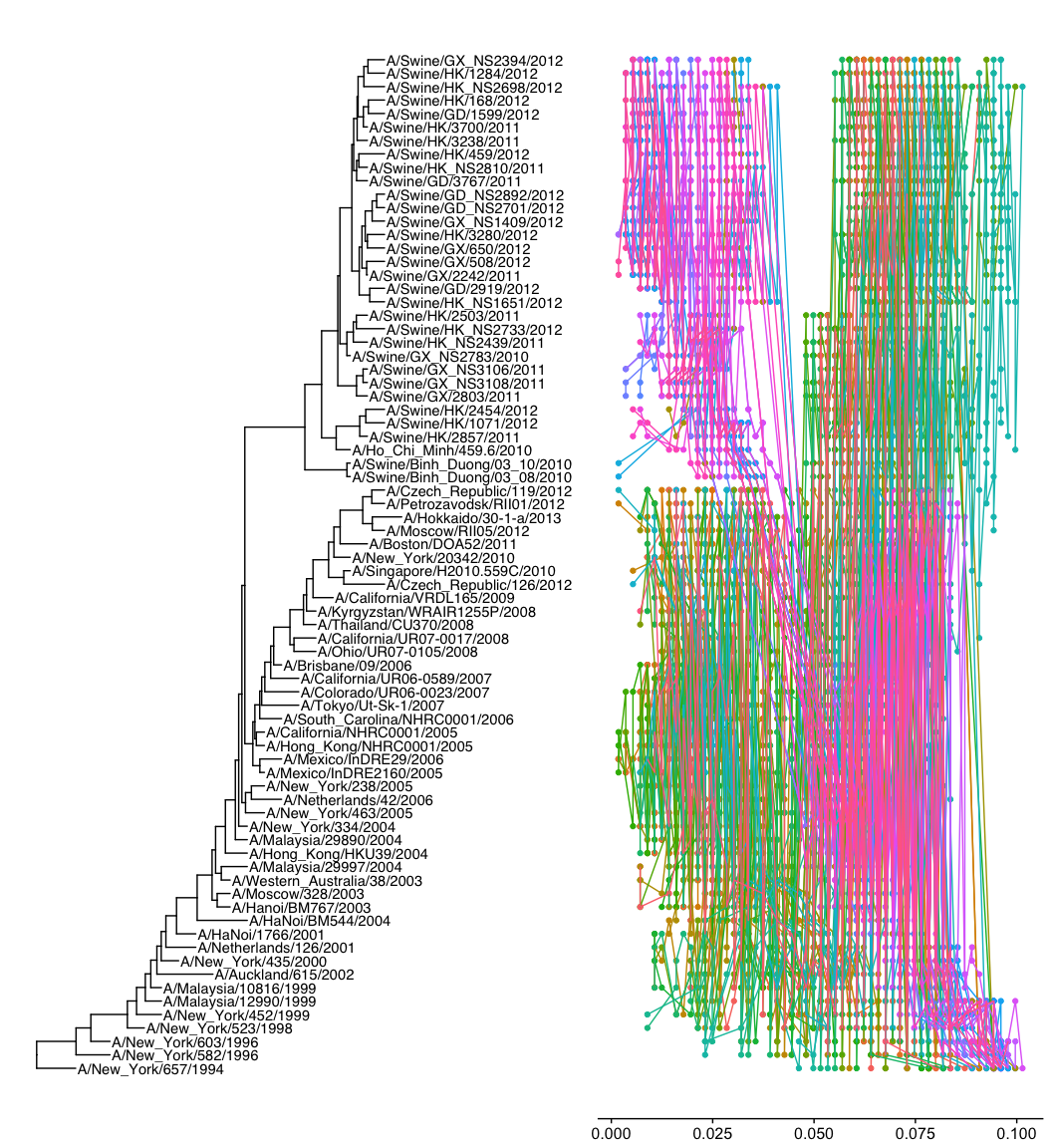
用散点可视化一个矩阵
当然是ggplot2画,才好和ggtree兼容,你首先问自己,抛开树,你能右边吗?本身你有个矩阵,row的顺序要跟着树来,你可以先不管,先随意,这是y轴。那x轴呢?你矩阵里的数字,打点你总该会的,而点连线,同样column的数字打出来的点,就连在一起,你先按这个思路画出来,再来问后续的。
这是我在「知识星球」中的回答,然而小伙伴表示还是不懂。那么我来详细讲解一下:
首先我们读入一个FASTA数据:
require(treeio)
fasta <- system.file("examples/FluA_H3_AA.fas", package="ggtree")
aa <- read.fasta(fasta)
数据用treeio包里的read.fasta去读这个氨基酸序列,它有个好处是你直接as.character(aa[[i]])就是个字母的向量,方便比较。
n = length(aa)
d = matrix(NA, ncol=n, nrow=n)
nm = labels(aa)
rownames(d) = colnames(d) = nm
for (i in seq_len(n)) {
for (j in seq_len(i)) {
d[i, j] = mean(as.character(aa[[i]]) != as.character(aa[[j]]))
d[j, i] = d[i, j]
}
}
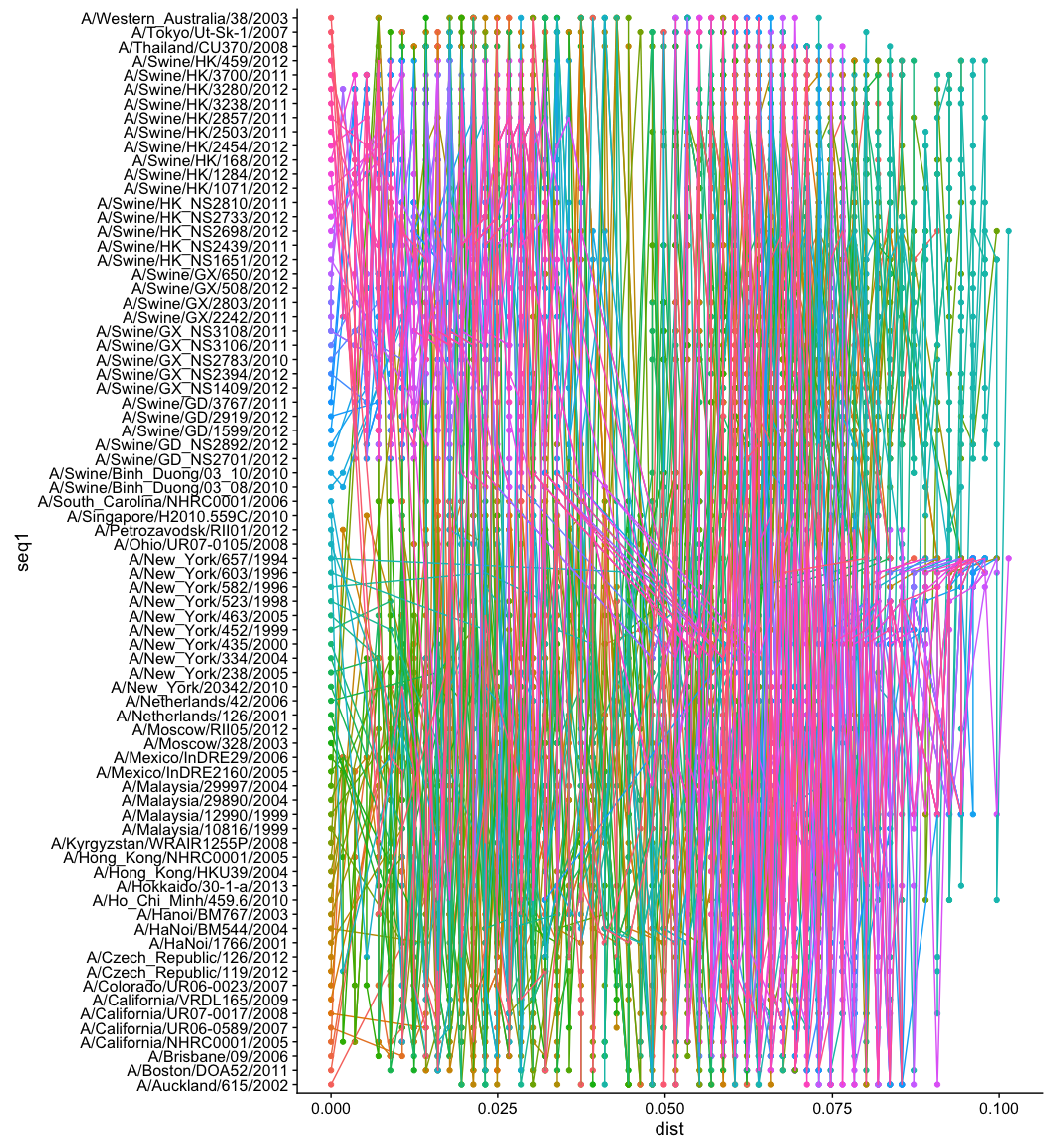
这段代码生成了pairwise distance矩阵,你可以搞其它的统计量,都OK的。大家可以想一想,一般这种矩阵怎么可视化?用热图!热图用数字来填充颜色,而现在我们把数字当成x轴上的变量,然后打点,仅此而已,我们有没有办法画出来,当然可以。同样row的数值会在同一y轴上,如果是热图,同一column的数值会在同一x轴上,方便横向和纵向比较,而现在同一column的数值不在同一直线一了,因为我们打的点的位置使用了矩阵中的数值,同一column的点被拉扯到不同的位置上去了,所以为了能够比较或者是看清楚同一column上的点的走势,我们把同一column上的点用线条连接在一起。这就是小伙伴提问的图,就是这么画出来的,非常简单。我在「知识星球」里其实已经解答得很清楚了。
画图实战
require(tibble)
dd <- as_data_frame(d)
dd$seq1 <- nm
require(tidyr)
td <- gather(dd,seq2, dist, -seq1)
这里宽变长,这种tidy格式适合于我们用ggplot2可视化。
> td
# A tibble: 5,776 x 3
seq1 seq2 dist
<fct> <chr> <dbl>
1 A/Swine/Binh_Duong/03_08/2010 A/Swine/Binh_Duong/03_08/2010 0.
2 A/Swine/Binh_Duong/03_10/2010 A/Swine/Binh_Duong/03_08/2010 0.00178
3 A/Swine/HK/2503/2011 A/Swine/Binh_Duong/03_08/2010 0.0267
4 A/Swine/HK_NS2439/2011 A/Swine/Binh_Duong/03_08/2010 0.0267
5 A/Swine/GX_NS2783/2010 A/Swine/Binh_Duong/03_08/2010 0.0249
6 A/Swine/GD/3767/2011 A/Swine/Binh_Duong/03_08/2010 0.0320
7 A/Swine/GX/2242/2011 A/Swine/Binh_Duong/03_08/2010 0.0338
8 A/Swine/GX/2803/2011 A/Swine/Binh_Duong/03_08/2010 0.0249
9 A/Swine/GX_NS3106/2011 A/Swine/Binh_Duong/03_08/2010 0.0285
10 A/Swine/GX_NS3108/2011 A/Swine/Binh_Duong/03_08/2010 0.0249
# ... with 5,766 more rows
看了上面的数据,看懂了我的解释,画图似乎是显而易见的。这里legend太多,所以用theme去掉不显示,实际操作中,比如提问者的图中,颜色没这么多,一般分clade来上色,或者如果你单纯想画这个图,你可以聚类,然后cut tree,通过子树来分类上色,或者使用kmeans聚类,直接上k种颜色。
require(ggplot2)
ggplot(td, aes(dist, seq1, group=seq2, color=seq2)) +
geom_point() + geom_line() +
theme(legend.position="none")
那么如何与树关联,首先我们要可视化相应的树,然后找出taxa在这颗树上的排列(可视化的角度,从上到下的顺序),我在ggtree里提供了get_taxa_name这个函数,方便用户得到这个排列,这样方便用户按照这个排列去做别的图,然后可以去拼图,这样可以保证图中的顺序与树是对应的。你可以用别的软件做图,你可以在AI里拼图,只要你能够按照顺序对应就行。
require(ggtree)
beast_file <- system.file("examples/MCC_FluA_H3.tree", package="ggtree")
beast_tree <- read.beast(beast_file)
p <- ggtree(beast_tree) + geom_tiplab() + xlim(NA, 30)
lv <- get_taxa_name(p)
td$seq1 = factor(td$seq1, levels=rev(lv))
我们排序后,重新画,会发现规律就出来了,其实你如果是单纯要画这个图,用聚类算法来排个序,也能出来挺好的效果,如果减少了颜色的数目,如我前面所说,规律应该更加清楚。当然我这个演示的,没有用户提问的图那么清楚漂亮,你知道原因吗?因为我这颗树的树型是梯子状的,而用户的那颗树是平衡的。可以说不看树,从这个矩阵点图,我们也可以大致猜出来树的形状!
p2 <- ggplot(td, aes(dist, as.numeric(seq1), group=seq2, color=seq2)) +
geom_point() + geom_line() + theme(legend.position="none")
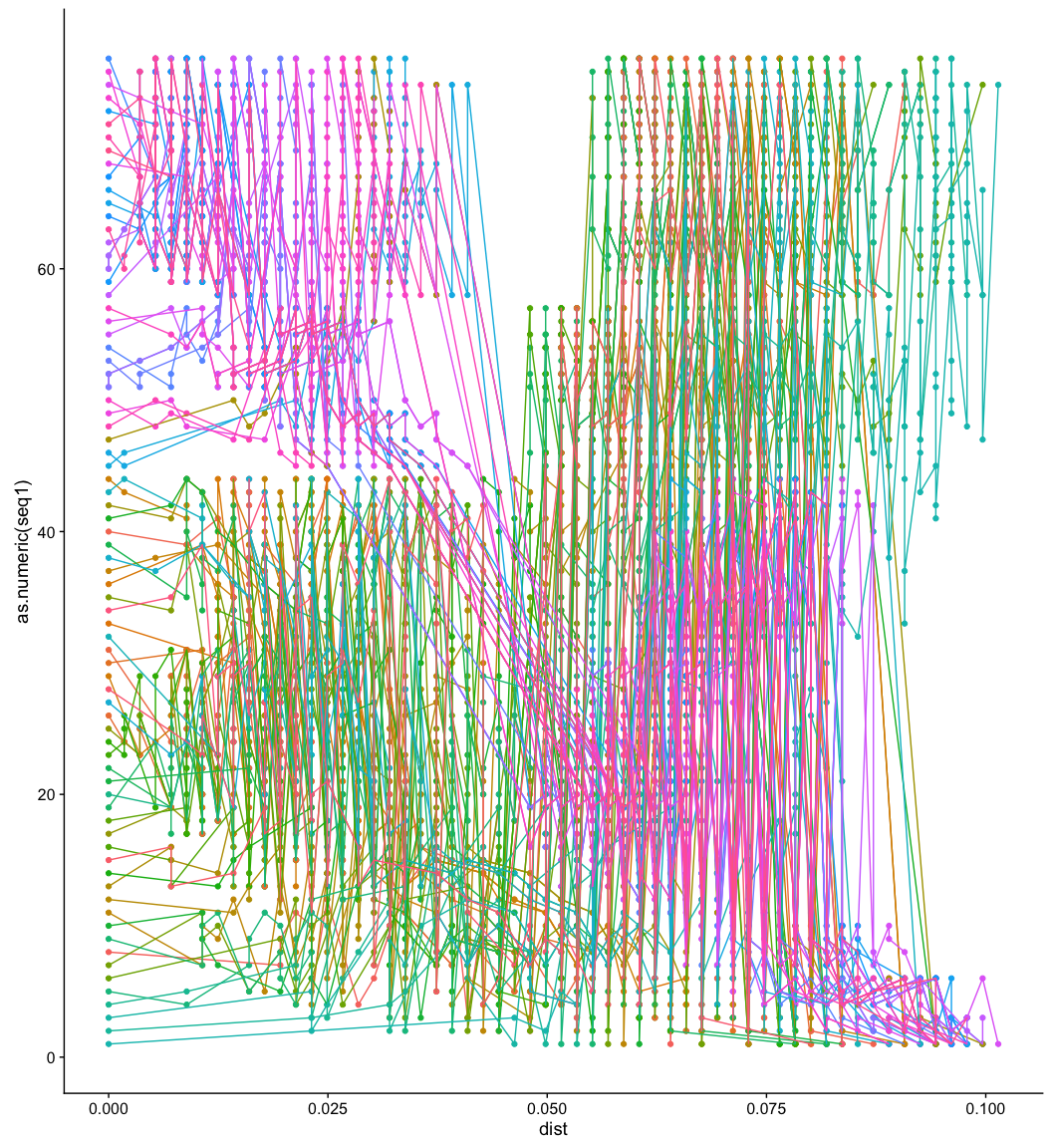
好了,最后是拼图,用cowplot,要使用align="h"参数,好对齐,ggtree画的图,y轴是数字型的,这也是为什么我在生成p2的时候,要使用as.numeric(seq1)把y轴变为数字型,大家都是数字型的,好对齐。
p2 <- p2 + theme(axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
axis.line.y=element_blank()) +
xlab(NULL) + ylab(NULL)
require(cowplot)
plot_grid(p, p2, align="h", rel_widths=c(1, .8))