2017眼看要结束,立下写《CS0: ChIPseq从入门到放弃》的flag还没完成,当时ChIPseeker是33个引用,现在已经80了,时间过得好快。
最近放羊的Jimmy给我发来了一个截屏:
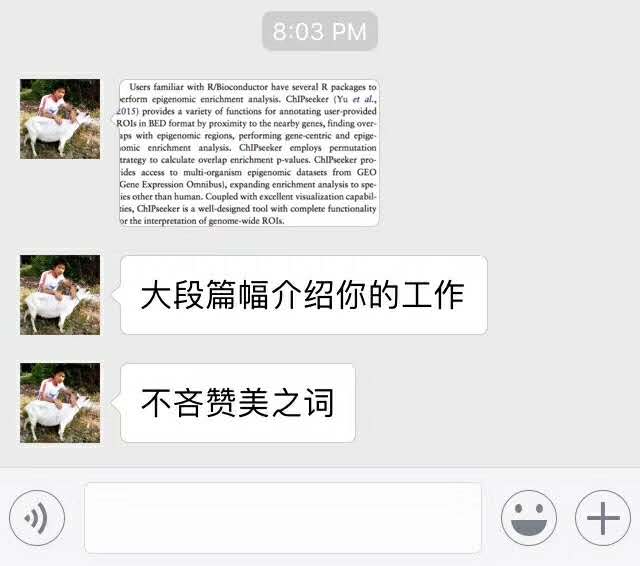
说了一篇新文章,大段在称赞ChIPseeker:
2017眼看要结束,立下写《CS0: ChIPseq从入门到放弃》的flag还没完成,当时ChIPseeker是33个引用,现在已经80了,时间过得好快。
最近放羊的Jimmy给我发来了一个截屏:

说了一篇新文章,大段在称赞ChIPseeker:
Since the clusterProfiler is a very useful tools for GO and Kegg annotation.At present I want to use it to enrich for kegg result while only have the KO number ,So I want to convert the KO number to the pathway function,Is there have any function or methods in the software can convert it?any help will be appreciated
这个问题问说他想转KO到通路,首先这是一个常见的错误,很多人分不清K和ko,所以在我告诉他可以把K number转成ko pathway的时候,我先指出他的错误。
ko is actually pathway map. I think you are talking about K number mapping to ko pathway.
> bitr_kegg("K00844", "kegg", "Path", "ko")
kegg Path
1 K00844 ko00010
2 K00844 ko00051
3 K00844 ko00052
4 K00844 ko00500
5 K00844 ko00520
6 K00844 ko00521
7 K00844 ko00524
8 K00844 ko01100
9 K00844 ko01110
10 K00844 ko01120
11 K00844 ko01130
12 K00844 ko01200
13 K00844 ko04066
14 K00844 ko04910
15 K00844 ko04930
16 K00844 ko04973
17 K00844 ko05230
微信公众号平台大部分人都是抄来抄去的二手资料,像joyplot这种特别新的东西,就很难看到,我试着搜索了一下,发现只有三篇,都是我写的。
在《你所不知道的,R的N种打开方式》一文中,我介绍了R的N种界面,这里将介绍一个用python写的极其现代化的R命令行界面,rtichoke(之前叫rice)之于R,就像ipython之于python一样。
最初github上用户(@JustGitting)报告说geom_hilight和geom_cladelabel不能用于unrooted树。详见:https://github.com/GuangchuangYu/ggtree/issues/118
我表示这确实是不行的,然后这就变成了feature request。我继续表示unrooted tree在ggtree中的实现并不好,我只实现了equal angle algorithm,在写注释图层之前,如果我有时间的话,我希望可以先实现更好的layout algorithm。
然后JustGitting表示,他发现python的ETE和R的ape,在无根树的可视化上好像都不太行。问我有什么unrooted的layout algorithm,有没有什么参考文献,或许他可以帮忙实现,因为他觉得ggtree是最成熟的软件。