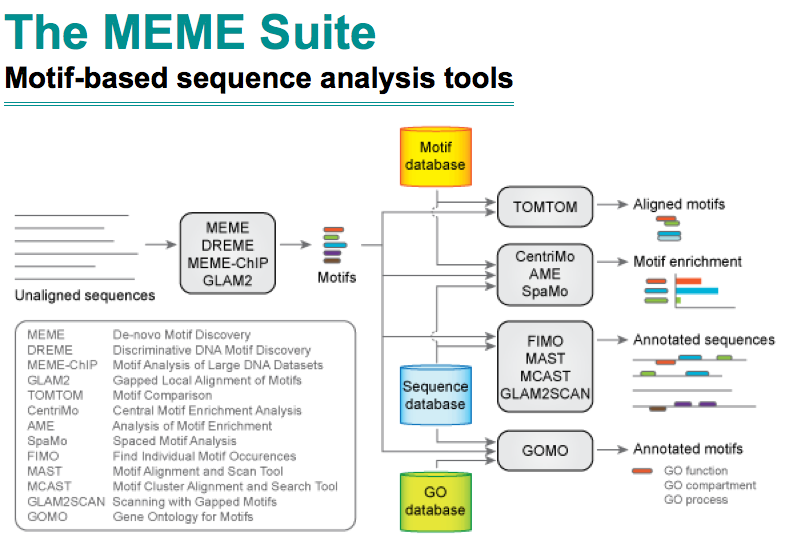
MEME是motif分析的webserver,所谓的注释序列就是一条线条上画一些长方形的box。
ppiPre抄袭了GOSemSim的代码,证据当然非常充分,比对一下代码就知道了,我在Proper use of GOSemSim一文中,做出了一些比较,另外也可以参考github页面,github记录了ppiPre被暴光抄袭之后所做的修改。 从我给BMC Systems Biology的编辑反馈这件事开始,在这铁板钉钉的事实面前,编辑拖了整整一年,而这一年时间过去了,ppiPre仍没有被编辑部受理。从最早反应这件事情,编辑信誓旦旦说他们很重视这种事情,到后面对我的邮件视而不见,我愿意相信编辑部处理这些事情,需要时间,他们有自己的规则,但一年的时间,不回邮件,冷处理以淡化此事,这绝对不是应该有的规则。 在编辑一直无视我的情况下,我写出了Proper use of GOSemSim一文,列举了一些一模一样的代码,并告知CRAN,当ppiPre被CRAN移除时,我写信给编辑,这时候,编辑告诉我说他们准备要去联系作者了,这时候已经过去半年了,是的!你没有看错,半年过去了,编辑说他们还没去联系作者!我是不相信的。必然是联系了之后,有某些不为人知的原因,所以编辑态度反常,对抄袭这种打鸡血的事情,不断在打太极。
再过二个月,ppiPre的作者邓岳给我写了信:
在进行测序的时候,需要将DNA打断,构建library,这些fragment需要接上adaptor,好进行扩增,illumina的测序,可以有single end和paired end两种,分别从一端和两端进行测序。
fragment ========================================
fragment + adaptors ~~~========================================~~~
SE read --------->
PE reads R1---------> <---------R2
unknown gap ....................